1. 学习目标
描述质量控制步骤后scRNA-seq分析的工作流程(聚类)。
现在有了高质量的细胞,可以继续工作流程。最终,希望对细胞进行聚类并识别不同的潜在细胞类型,但是在那之前需要完成几个步骤。下面的工作流程示意图中的绿色框对应于QC 后采取的步骤,共同构成了聚类工作流程。
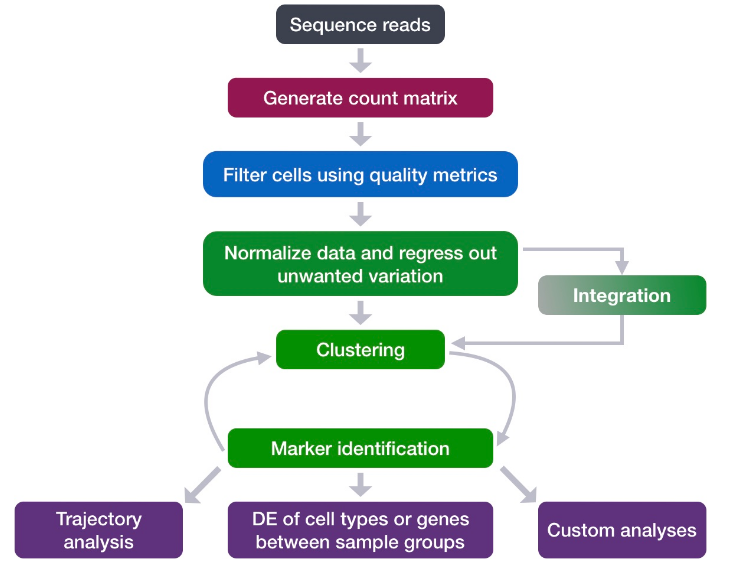
2. 聚类流程
对于具有信息性的事物,它需要表现出变化,但并非所有变化都是信息性的。聚类分析的目的是保留数据集中应该定义的细胞类型的主要变异来源,同时消减由于无意义的变异来源(测序深度、细胞周期差异、线粒体表达、批次效应等)引起的变异。然后,为了确定存在的细胞类型,将使用特异的基因进行聚类分析,以定义数据集的主要来源。
要识别clusters,将执行以下步骤:
2.1. 变异溯源
- 探索
unwanted variation的来源
工作流程的第一步是查看数据是否包含任何不需要的变异。在scRNA-seq数据中评估的最常见的生物学效应是细胞周期对转录组的影响。另一种已知的生物学效应是线粒体基因表达,它被解释为细胞应激的一种迹象。工作流程的这一步涉及探索数据以确定想要回归的协变量。
2.2. 回归
- 归一化和回归
unwanted variation来源
Seurat最近推出了一种名为 sctransform的新方法,该方法对 scRNA-seq数据执行多个处理步骤。需要标准化以缩放原始计数数据以获得细胞之间正确的相对基因表达丰度。sctransform函数实现了数据的高级归一化和方差稳定。 sctransform函数还回归了数据中不需要的变化的来源。在上一步中,已经确定了这些可变性来源,在这里指定了这些协变量是什么。
2.3. 整合
- 整合(可选)
通常使用 scRNA-seq,会处理对应于不同样本组、多个实验或不同模式的多个样本。如果想最终比较组之间的细胞类型表达,建议整合数据。整合是一种强大的方法,它使用这些最大变化的共享源来识别跨条件或数据集的共享子群。在Seurat中执行整合涉及几个步骤。完成后,使用可视化方法来确保良好的整合,然后再对单元进行聚类。
2.4. 细胞聚类
- 细胞聚类
通过基于其基因表达谱的相似性对细胞进行分组来获得细胞簇。表达轮廓相似性是通过距离度量来确定的,它通常以降维表示作为输入。Seurat 根据从整合的最多变基因的表达中获得的 PCA分数将细胞分配到簇中。
2.5. 质量评估
- 聚类质量评估
数据中确定的clusters代表可能属于相似细胞类型的细胞组。在确认一组成员单元格的单元格类型之前,需要执行以下步骤:
- 检查
clusters是否不受无意义变异来源的影响。 - 检查主要的主成分是否在驱动不同的
clusters。 - 通过查看
clusters中已知标记的表达式来探索细胞类型身份。
本文由mdnice多平台发布
原文地址:http://www.cnblogs.com/swindler/p/16864093.html
